ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- HOME
- ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- 製品・サービス詳細
- 新しいMagMAXキットとKingFisherプラットフォームで自動化したヒトマイクロバイオームのメタゲノム解析
会社カテゴリー:研究関連資材
主サービス提供地域:
製品・サービス詳細
新しいMagMAXキットとKingFisherプラットフォームで
自動化したヒトマイクロバイオームのメタゲノム解析
概要
• Applied Biosystems™ MagMAX™ Microbiome Ultra NucleicAcid Isolation Kitは、ヒト糞便などのさまざまな生体サンプルから高品質かつ阻害物質フリーのDNAおよびRNAを抽出します。
• 抽出した核酸は、試験サンプルの包括的な遺伝子プロファイリングに不可欠なメタゲノミクスシーケンシングなどの各種技術に適した品質を誇ります。
• MagMAX Microbiome Ultra Nucleic Acid Isolation KitとThermo Scientific™ KingFisher™ 核酸・タンパク質・細胞自動抽出・精製装置の併用が全体処理時間を短縮し、迅速なワークフローを実現します。
はじめに
ヒト微生物叢(マイクロバイオーム)は、さまざまな疾患の診断・治療に重要な役割を果たす存在として近年注目を浴びています。マイクロバイオームを構成する微生物の適切な同定と存在量(アバンダンス)の算出が大きな壁となってはいますが、メタゲノミクスシーケンシング技術の進歩もあり、試験サンプルに含まれる全ての遺伝子の包括的なゲノムプロファイルを解析できるようになりました。
ヒトマイクロバイオームプロジェクト(HMP)の研究成果発表によると、個々の宿主や部位ごとに優勢となるタクソンが異なることが明らかになりました。また、患者から得たメタゲノムデータと健康被験者からのメタゲノムデータを比較することで、さまざまな疾患の発現や予防に関与する細菌が数多く特定されました[1]。
ヒト腸は、内部に大量のマイクロバイオータ(細菌叢)が共生しており、研究が最も深く進んでいるエコシステムの1つでもあります[2]。これらの解析には、採取しやすく大量のバイオマスを含有する糞便サンプルの利用が一般的です。糞便ほど研究されてはいないものの、唾液、尿等の体液にもそれぞれ固有の細菌叢(バクテリオーム)が存在しています。
宿主や各部位のマイクロバイオームのプロファイリングには、微生物からの DNA 抽出が解析前の重要な鍵となりますが、マイクロバイオーム構成菌の正確なゲノム解析に十分な量のDNA 精製や、タイプの異なるサンプル間での解析結果の同等・比較性(コンパラビリティ)には課題が残ります。本資料では、糞便、尿、唾液からのトータル核酸の抽出にいかんなく発揮されるApplied Biosystems™ MagMAX™ Microbiome Ultra Nucleic Acid Isolation Kit の性能をご紹介します。各サンプルにおけるマイクロバイオームの多様性をメタゲノミクスシーケンシングで細かく検証したこの試験は、疾患バイオマーカーとしてのマイクロバイオーム利用の可能性に光をあてるものといえます。
材料・方法
2 名の健康ドナーから糞便、唾液、尿サンプルを採取し、同日中にMagMAX Microbiome Ultra Nucleic Acid Isolation Kit を用いてトータル核酸を 2 回ずつ独立して抽出しました。糞便 100 mg、唾液 400 µL、尿 400 µL のインプットから、Thermo Scientific™ KingFisher™ Flexシステム(96ディープウェルヘッド)でトータル核酸を自動抽出し、Thermo Scientific ™ NanoDrop™ 微量分光光度計で抽出量を計測しました。糞便から抽出したトータル核酸調製物の純度と品質を、分光光度計で計測した吸光度(A260/A280)と1%アガロースゲル電気泳動を基に評価しました。
次に、Applied Biosystems ™ TaqMan ™ AssaysとApplied Biosystems ™ TaqMan ™ Fast Advanced Master Mixを用いて、高速サイクリング条件下で各サンプルの qPCR 解析(triplicate)を行いました。各反応には核酸調製物の 20%(容量%)を用いました。
メタゲノミクス処理
ZymoBIOMICS™ Shotgun Metagenomic Sequencing Serviceによるメタゲノミクスシーケンシングのために、糞便、唾液、尿から抽出したトータル核酸サンプルをZymo Research(カリフォルニア州アーバイン)へ送付しました。
業者指定プロトコルどおりにKAPA™ HyperPlus Kitを用いて、100 ng の DNAインプットからシーケンシングライブラリーを調製しました。ライブラリーには内因性 8 bp バーコード配列とIllumina™ TruSeq™ アダプター配列を付加しました。全てのライブラリーをAgilent ™ TapeStation ™ システムで定量して等量をプールした後、最終プールをqPCR で定量し、Illumina ™ HiSeq™ システムでシーケンスしました。
生のシーケンスリードデータから、Trimmomatic-0.33を用いて質の低い断片とアダプター配列をトリミングしました[3]。クオリティトリミングの設定はスライディングウィンドウを6 bp、クオリティカットオフを20し、70 bp未満のリードを取り除きました。構成微生物のプロファイリングにはMetaPhlAn2[4]を、視覚化にはKronaTools[5]を用い、MetaPhlAn2出力データから菌種レベルでの存在量情報を抽出し、以下の追加解析を行いました。
• 構成微生物のバープロット作成と、QIIME[6]を用いた(Bray-Curtis非類似度に基づく)β多様性解析
• 独自のインハウスPython™ スクリプトを用いた(Bray-Curtis非類似度に基づく)階層クラスター解析を基に、分類群のアバンダンスを示したヒートマップを作成
• デフォルト設定(p>0.05、LDA effect size >2)でのLEfSeバイオマーカーディスカバリー解析[7]
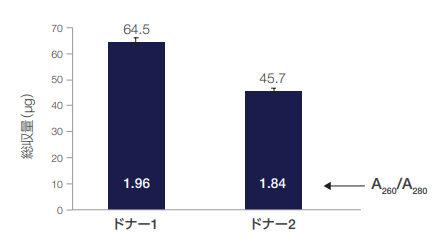
図 1.糞便サンプル 100 mgから抽出したトータル核酸量
ドナー2 名から採取したサンプルをMagMAX Microbiomeキットを用いて処理しました。96ディープウェルヘッドのKingFisher Flexシステムを用いて、ビーズ式ホモジナイザーによる細菌破砕ステップを加えたプロトコルで核酸を抽出しました。グラフ中の数字はA260/A280です。
結果
トータル核酸 - 抽出量、純度、品質
MagMAX Microbiome Ultra Nucleic Acid Isolation Kitを用いたハイスループット抽出法は、革新的な磁気ビーズベースのケミストリーとビーズ式ホモジナイザー(細菌破砕ステップ)によって迅速なサンプル処理が可能となり、Thermo Scientific ™ KingFisher™ FlexシステムやThermo Scientific™ KingFisher™ Duo Primeシステムを用いて最大96サンプルを同時精製できます。糞便サンプルでは100 mgのインプットから高収量(45~64 µg、図1)のトータル核酸を抽出でき、唾液および尿サンプルでは400 µL のインプットからそれぞれ1.5~2.5 µgおよび0.2~0.3 µgのトータル核酸を抽出できました(データ未掲載)。抽出したトータル核酸のA260/A280はサンプルタイプにかかわらずいずれも1.7を上回り、調製物の純度の高さを確認できました。糞便サンプルから精製したトータル核酸のアガロースゲル電気泳動からは、高品質の細菌由来DNAおよびRNAを回収できたことが示されました(図2)。

図 2.糞便から精製した微生物由来トータル核酸の品質
ドナーごとに2回ずつ抽出したトータル核酸サンプル各1 µgを分取して1%アガロースゲルで電気泳動した結果、ゲノムDNA および RNAともに明瞭なバンドを検出できました。断片サイズの確認にはInvitrogen ™ 1 Kb Plus DNA Ladderを用いました。変性アガロースゲルではないので、測定した見かけのRNAサイズは実際のサイズとは異なります。
糞便、唾液、尿サンプルから抽出したトータル核酸を用いて、ファーミキューテス門(グラム陽性細菌門)、バクテロイデーテス門(グラム陰性細菌門)、E. coli(大腸菌)に対するTaqMan qPCR 解析を実施しました。いずれも常在菌として体内から検出される菌ですが、通常、E. coli 量は多くはありません。上記2種類の細菌門とE. coliを調べることで、広範囲の細菌をカバーできました。結果は、想定通り、糞便サンプルにはファーミキューテス門とバクテロイデーテス門の細菌群が多く(図3A)、尿サンプルと唾液サンプルにはファーミキューテス門の細菌群が豊富でした(図3B、図3C)。大腸菌の存在量は低かったものの、どの試験サンプルも検出範囲に収まっていました(図 3A、図3B、図3C)。Applied Biosystems ™ Xeno™ DNAコントロールとRNAコントロールを用いたTaqMan Assayではトータル核酸調製物による阻害は認められず(データ未掲載)、MagMAX Microbiome Ultra Nucleic Acid Isolation Kitを用いることで、最も扱いの難しいサンプルからも高品質かつ阻害物質フリーの核酸を抽出できることが確認できました。
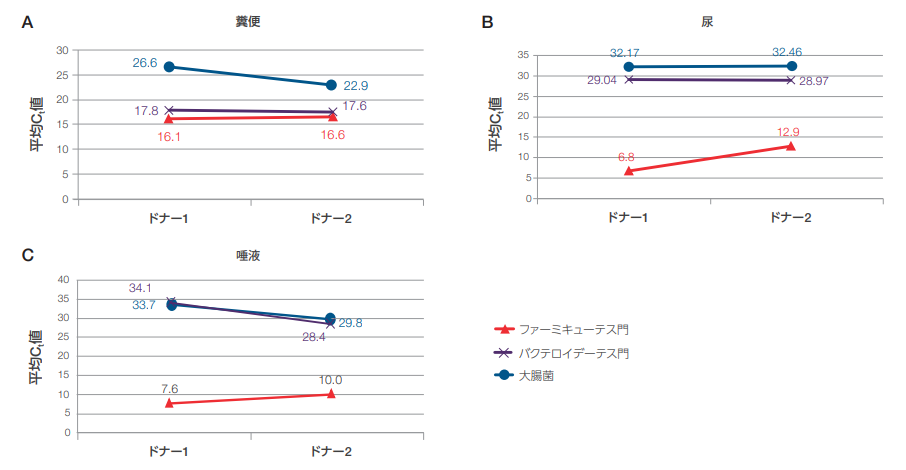
図3.ドナー2名の(A)糞便、(B)尿、(C)唾液サンプルからMagMAX Microbiome Ultra Nucleic Acid Isolation Kitを用いて精製したDNAのqPCR解析
グラム陽性菌1カテゴリー(ファーミキューテス門)とグラム陰性菌2カテゴリー(バクテロイデーテス門、大腸菌)についてTaqMan Assayを行いました。ファーミキューテス門とバクテロイデーテス門の解析にはトータル核酸サンプルを1:100に希釈して用い、大腸菌の解析には未希釈のインプットを用いました。高速サイクル条件下でTaqMan Fast Advanced Master Mixを加えて反応させました。
糞便、尿、唾液から抽出したトータル核酸のショットガンメタゲノミクスシーケンシング
ドナー2名の糞便、尿、唾液サンプルからトータル核酸を抽出してシーケンスし、全サンプルのメタゲノミクスシーケンシングデータを存在量ヒートマップとして図 4に示します。この存在量ヒートマップは、タイプの異なる各サンプルに含まれていたマイクロバイオーム全てを網羅したプロファイルではありません。菌種レベルでの全ゲノムプロファイルは図 5に示します。なお、MagMAX Microbiome Ultra Nucleic Acid Isolation Kitは図 4 の尿・唾液中プロファイルにみられるように細菌だけでなくウイルス由来のトータル核酸も抽出できます。糞便と唾液のメタゲノミクスプロファイルに共通するターゲットはPrevotella種の細菌のみでしたが、尿と唾液のメタゲノミクスプロファイルには共通のウイルスターゲットが認められました。糞便から抽出した核酸のメタゲノミクスシーケンシングの結果、ドナー1の糞便サンプル(zr2132.1)はPrevotella、Faecalibacterium、Roseburia、Bacteroides、Alistipes等が豊富で、ドナー2の糞便サンプル(zr2132.2)は複数のBacteroides、Faecalibacterium、Bifidobacterium、Parabacteroidesなどが豊富に認められました。Centrifuge プログラムを用いて全サンプルをヒトデータベースと照らし合わせてマッピングしたところ(データ未掲載)、MagMAX Microbiome Ultra Nucleic Acid Isolation Kitは最大99%の細菌ターゲットの抽出に性能を発揮し、ヒト糞便サンプル中の極めて微量(0.06~0.45%)のターゲットも検出可能であることを示しました。
菌種レベルでの全遺伝子のメタゲノムプロファイルからは、各サンプルタイプ固有の菌叢プロファイルに含まれるさまざまな細菌ターゲットとウイルスターゲットが見て取れます。両ドナーの糞便サンプルにはバクテロイデーテス門、ファーミキューテス門、放線菌門、プロテオバクテリア門の細菌群が豊富に認められており、ドナー1 の糞便サンプルはPrevotella copri、複数の Bacteroides 種、Rothia、Faecalibacterium、Sutterella など、ドナー2 の糞便サンプルは Bacteroides、Bifidobacterium、Faecalibacterium、Rothia、Sutterella などが豊富でした。また、両ドナーともに、尿サンプルではLactobacillus、Streptococcus、Bacteroides、Ruminococcus、Bifidobacterium、Pseudomonasファージなど、唾液サンプルでは Streptococcus mitis、その他の Streptococcus 種、Granulicatella、Neisseria、Prevotellaなどが豊富でした(図5)。
結論
新しいMagMAX Microbiome Ultra Nucleic Acid Isolation Kitを用いてヒト糞便などのさまざまな生体サンプルから高品質かつ阻害物質フリーのDNAおよびRNAを抽出する迅速自動ワークフローを構築し、ドナー2 名からの採取サンプルを対象に全ゲノムシーケンシングを行ったところ、糞便、尿、唾液に存在する全ての微生物プロファイルを示すことができました。ヒト体内に存在する細菌叢は、特定疾患の診断的バイオマーカーとして活用できるのに加え、将来的にマイクロバイオームをベースとした治療の道を開く可能性も秘めています。マイクロバイオーム研究の力を最大限に活かすために開発した今回のワークフローは、微生物叢に関するメタゲノムデータの迅速な作成を可能にします。
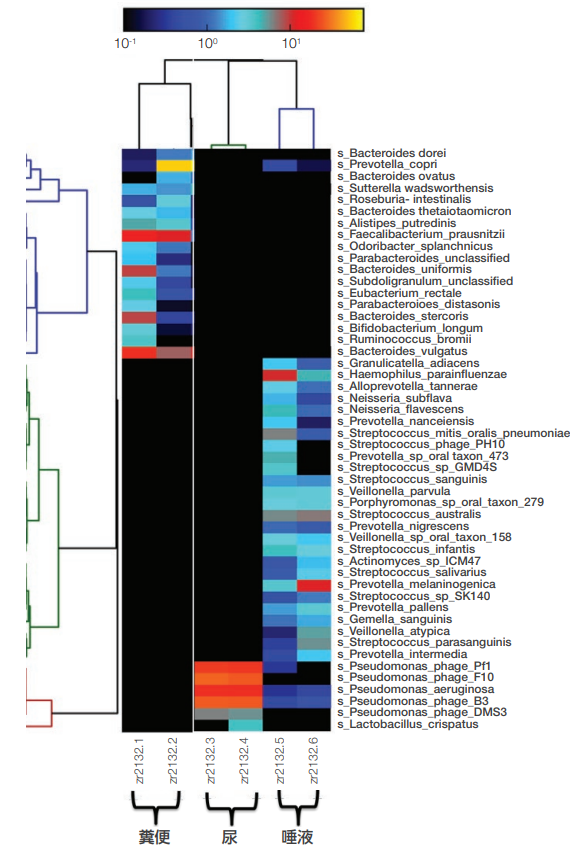
図4.ショットガンメタゲノミクスシーケンシングによるアバンダンス・ヒートマップ(糞便、尿、唾液サンプル)
菌種レベルでのショットガンシーケンシングデータ
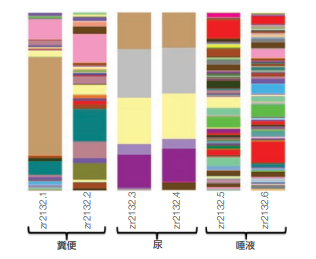
図 5.ショットガンメタゲノムシーケンシングによる全遺伝子のメタゲノムプロファイル(糞便、尿、唾液サンプル)
サンプルごとに同定された操作分類単位(Operational Taxonomic Unit:OTU)で色分けしています。
菌種レベルでのショットガンシーケンシングデータ

参考文献
1. Patrice CD (2018) Human gut microbiome: hopes, threats and promises. Gut 67:1716–1725.
2. Conrad R, Vlassov A (2015) The human microbiota: composition, functions, and therapeutic potential. Med Sci Rev 2:92–103.
3. Bolger AM, Lohse M, Usadel B (2014) Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics 30:2114–2120.
4. Segata N, Waldron L, Ballarini A et al. (2012) Metagenomic microbial community profiling using unique clade-specific marker genes. Nat
Methods 9:811–814.
5. Ondov BD, Bergman NH, Phillippy AM (2011) Interactive metagenomic visualization in a Web browser. Bioinformatics 12:385.
6. Caporaso JG, Kuczynski J, Stombaugh J et al. (2010) QIIME allows analysis of high-throughput community sequencing data. Nat Methods
7:335–336.
7. Segata N, Izard J, Waldron L et al. (2011) Metagenomic biomarker discovery and explanation. Genome Biol 12:R60.