ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- HOME
- ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- 製品・サービス詳細
- 2つのマグネットヘッドとグレードアップされた冷却、加温および溶出機能を搭載したKingFisher Apexシステム
会社カテゴリー:研究関連資材
主サービス提供地域:
製品・サービス詳細
2つのマグネットヘッドとグレードアップされた冷却、
加温および溶出機能を搭載したKingFisher Apexシステム
高いプロセス効率と最適なサンプル抽出の達成
主要な特長
- Thermo Scientific™ KingFisher™ Apexシステムは、2つのマグネットヘッドをセットできるオプションによって、マグネットヘッドの交換による中断なしにさまざまなキットやプロトコルを使用する複数の抽出を連続的に実行できます。これにより多様な種類のサンプルを短時間に処理することが要求されるラボで、時間とリソースの節約に貢献できます。
- KingFisher Apexシステムのグレードアップした冷却および加温機能と、ストレージチューブへの直接溶出により、プロセスの効率化と抽出したサンプルの品質が改善されます。サンプルは抽出工程が全て管理された環境中に保持されるため、サンプルの品質と再現性が維持されます。ストレージチューブへの溶出により、DNAおよびRNAサンプルの簡単で効率的かつ合理化された長期保存が可能となり、磁気ビーズベースの抽出による溶出サンプルの長期間のバイオバンキングやダウンストリームのアプリケーションへの移行がしやすくなります。
- KingFisher Apexシステムに新たに搭載されたUVランプ機能は装置内の滅菌を可能にします。
- RNA、DNA の抽出および細胞の分離に関して KingFisherApexシステムは先行の Thermo Scientific ™ KingFisher ™Flexシステムと同等の収量、容量および純度を提供します。
- ワークフローの改善のため、バーコード付きの最適化されたプラスチック消耗品が用意されています。
はじめに
磁気ビーズをベースとする分離技術は、RNA、DNA および細胞分離のための重要な技術の 1つです。さまざまな磁気ベースのキットを有するThermo Scientific™ KingFisher ™シリーズは、この技術を使用して高純度で高品質な抽出を達成し、一貫性のある結果の入手を可能にします。KingFisher Flexシステムは世界中の多くの診断および研究ラボにおいて、さまざまなApplied Biosystems™ MagMAX™抽出キットを用いた、ハイスループット抽出に使用されてきました。近年のSARS-CoV-2感染の世界的危機の状況下では、ウイルス輸送培地(VTM)中の鼻咽頭スワブおよび唾液サンプルからの SARS-CoV-2ウイルス RNA 抽出の大規模なハイスループット処理のためにKingFisher FlexシステムがApplied Biosystems™ MagMAX™Viral/Pathogen Kitとともに使用されています。
KingFisher Apexシステムは、KingFisher Flexシステムの優れた機能に基づいて構築されており、磁気ビーズベースのハイスループット技術で期待される高品質の結果を提供します。クラウドベース技術の採用およびタッチスクリーンによるユーザーインターフェースの改善に加えて、KingFisher Apexシステムには、サンプル冷却、2つのマグネットヘッド間の切り替え、ストレージチューブへの直接溶出および UV ランプによる滅菌といった新たな機能が搭載されています。これらの機能の有効性をご紹介するために、KingFisher ApexシステムおよびKingFisher FlexシステムとApplied Biosystems™ MagMAX™抽出キットシリーズを使用し、全血、生体液、バフィーコート、口腔粘膜スワブ、血漿および組織などさまざまな種類のサンプルからRNA、DNA の抽出を行い、両システム間の抽出効率および核酸の品質、収量および純度の解析・比較を行いました。
磁気ビーズをベースとした MagMAXシリーズを用いた核酸抽出
材料および方法
KingFisher ApexシステムとKingFisher Flexシステムが同等な性能であることを確認するために、5 種類の Applied Biosystems ™ MagMAX ™キットを使用して、KingFisher ApexシステムおよびKingFisher Flexシステムを用い、さまざまなタイプのサンプル、ワークフローおよびスタートサンプル量を設定して核酸抽出を行いました(図1)。

図1. KingFisher FlexおよびKingFisher Apexシステムの比較テストに使用した MagMAX キット。
Applied Biosystems ™ MagMAX ™ DNA Multi-Sample Ultra 2.0 Kit、MagMAX ™ mirVana ™ Total RNA Isolation Kit、MagMAX ™ Cell-Free DNA Isolation Kit、MagMAX ™
Cell-Free TNA Isolation KitおよびMagMAX™ FFPE DNA/RNA Ultra Kitを用い、KingFisher ApexおよびKingFisher Flexの両システムで抽出を行いました。
KingFisher Apexシステムおよび KingFisher Flexシステムを使用した実験は、全て並行して行いました。本試験では、Combi チップコームとディープウェルプレート(96または 24ウェル)を使用して KingFisher Apexシステムの機能を評価しました。ホルマリン固定パラフィン包埋(FFPE)サンプルからの核酸(DNA/RNA)抽出の比較では、皮膚、肺、結腸、乳房組織の FFPE サンプルを用い、MagMAX FFPE DNA/RNA Ultra Kit のスタンダード用と大容量用の両プロトコルを用いて処理しました。ゲノム DNA 抽出の比較では、ヒト全血およびバフィーコートサンプルを用い、サンプルのインプット容量を振り(200 µL、400 µL、1 mLおよび2 mL)、MagMAX DNA Multi-Sample Ultra 2.0 Kitを使用して処理しました。RNA抽出の比較ではMagMAX mirVana Kitを用い、全血サンプルは生体液プロトコルで処理し、培養細胞サンプル(HeLa 細胞および Huh7 細胞)は、インプット容量を振って細胞・組織プロトコルを使用して処理しました。セルフリー核酸(DNA/RNA)の回収比較では、MagMAX Cell-Free DNA Isolation KitおよびMagMAX Cell-Free TNA Isolation Kitを使用し、血漿サンプル(2 mLおよび4 mL)のプロトコルを使用して処理しました。
RNA収量の確認にはInvitrogen™ Qubit™ RNA HS Assayを、DNA収量の確認にはInvitrogen™ Qubit™ dsDNA HS Assayを使用して、Invitrogen ™ Qubit™ 4.0 Fluorometerでそれぞれ解析を行いました。核酸サンプルの純度は、Thermo Scientific™ NanoDrop ™ 8000微量分光光度計を使用し、A260/A230およびA260/A280の吸光度比の値で判断しました。ゲノム DNA(gDNA)、mRNA およびセルフリーDNA(以後cfDNA)の品質の評価には、Agilent ™ 2100バイオアナライザを使用し、回収した核酸のピークサイズを解析しました。またqPCRを用いた評価ではApplied Biosystems™ QuantStudio™ 12K Flexシステムを用い、適切な反応試薬を使用してmRNA、miRNAまたはDNAの発現解析を行いました。
結果および考察
MagMAX FFPE DNA/RNA Ultra Kitを用いた核酸抽出結果の比較
MagMAX FFPE DNA/RNA Ultra Kitを使用し、スタンダードプロトコルで、乳房、結腸および皮膚の腫瘍のFFPEサンプルからの抽出を行いました。大容量用プロトコルで、皮膚、肺および乳房の腫瘍のFFPEサンプルからのDNAとRNAの抽出を行いました。図2に、KingFisher FlexおよびKingFisher Apexの両システムから、各プロトコルを使用して得られたRNAおよびDNA の収量の詳細を示します。3 種類のサンプル全てにおいてほぼ同等の収量が得られ、標準偏差内の収量が達成されていることが分かります。
qPCRを利用した解析にはQuantStudio 12K Flexシステムを用い、qPCR標的(GAPDH_g 1)を使用しDNAの収量を測定しました。RNAはmRNA 標的(GAPDH_m1およびβアクチンACTB_m1)を使用して解析しました。
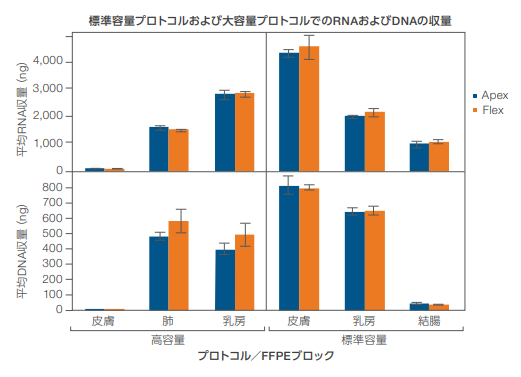
図2. KingFisher FlexおよびKingFisher Apexシステムを用いたスタンダードプロトコルおよび大容量プロトコルでの RNAおよび DNAの収量。
MagMAX FFPE DNA/RNA Ultra Kitを使用して、スタンダードプロトコルで乳房、皮膚、および結腸の FFPEサンプルから、大容量プロトコルで乳房、肺および皮膚の FFPEサンプルからそれぞれ抽出を行いました。DNAおよびRNAの収量はそれぞれ適切なQubitアッセイを使用して測定しました。
図3は、MagMAX FFPE DNA/RNA Ultra Kit のスタンダードプロトコルを使用して、皮膚、結腸および乳房腫瘍の FFPEサンプルから抽出した核酸(DNA/RNA)をqPCRで解析した結果です。KingFisher FlexおよびKingFisher Apex両システムから抽出した核酸間の Ct 値の差はそれぞれ1サイクル範囲内にあり、2つのシステムの核酸抽出が同等であったことを示しています。
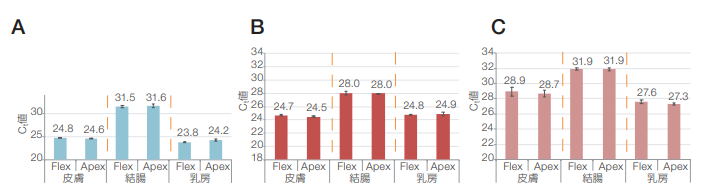
図3. スタンダードプロトコルで処理した腫瘍FFPE由来の抽出サンプルを用いたDNAおよびmRNA標的のqPCRデータ。
KingFisher FlexおよびKingFisher Apexシステムのスタンダードプロトコルを使用して処理した乳房、結腸および皮膚の腫瘍FFPEサンプル由来の抽出物を用いた(A)GAPDH DNA(、B)GAPDH_m1 RNAおよび(C)ACTB_m1 RNAのCt値。
図4は、大容量プロトコルを使用した、皮膚、肺および乳房の腫瘍サンプルに関する、DNA 標的(GAPDH_g1)ならびにmRNA標的(GAPDH_m1およびACTB_m1)の qPCR データを示しています。KingFisher FlexおよびKingFisher Apexシステムからの DNA および mRNA の収量が同等であったことを示しています。
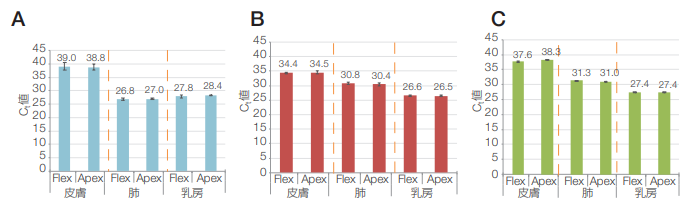
図4. 大容量プロトコルで処理した腫瘍FFPE由来の抽出サンプルを用いたDNAおよびmRNA標的のqPCRデータ。
KingFisher FlexおよびKingFisher Apexシステムの大容量プロトコルを使用して処理した皮膚、肺および乳房の腫瘍FFPEサンプル由来の抽出物を用いた(A)GAPDH DNA、(B)GAPDH_m1 RNAおよび(C)ACTB_m1 RNAのCt値。
両システム間での RNA サイズのプロファイルを、Agilent™バイオアナライザとAgilent™ High Sensitivity (HS) DNA Chipを使用して確認しました。図 5 および図6は、スタンダードおよび大容量プロトコルでKingFisher Flexおよび KingFisher Apexシステムを使用して抽出した腫瘍のFFPEサンプルのRNAサイズプロファイルを示しており、いずれのサンプルにおいても両システム間で同等であることを示しています。
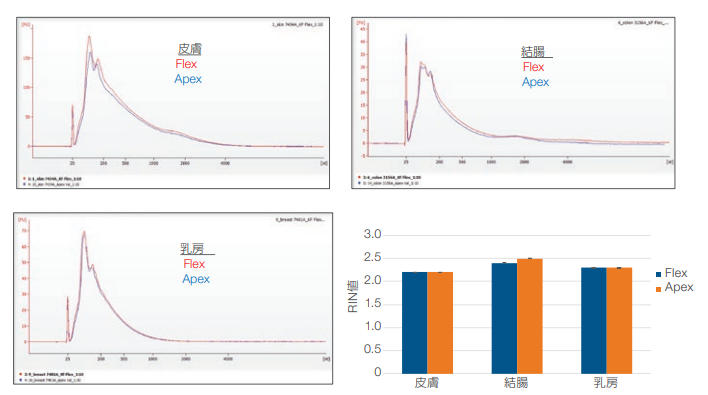
図 5. スタンダードプロトコルを使用して抽出したサンプルの RNA サイズプロファイルおよびRIN値。
Agilentバイオアナライザで解析した結果、KingFisher FlexおよびKingFisher Apexシステムを使用してスタンダードプロトコルで抽出したサンプルのRNAサイズプロファイルは一致していました。
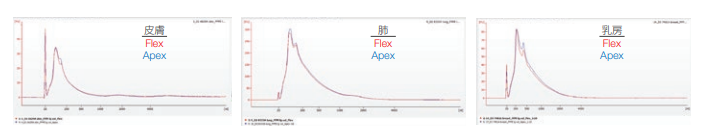
図6. 大容量プロトコルを使用して抽出したサンプルのRNAサイズプロファイルおよびRIN値。
Agilentバイオアナライザで解析した結果、KingFisher FlexおよびKingFisher Apexシステムで大容量プロトコルで抽出したサンプルのRNAサイズプロファイルは一致していました。
miRNA の検出テストでは、let7e および mir-16 を標的とした qPCR アッセイで解析を行いました。スタンダードおよび大容量のプロトコルで抽出した腫瘍の FFPE 由来の抽出サンプルを使用し、Applied Biosystems™ TaqMan® microRNA Reverse Transcription Kitで逆転写反応後、QuantStudio 12K Flexシステムで Applied Biosystems ™TaqMan® Universal Master Mixを使用したランを実行しました。
図7は、KingFisher FlexおよびKingFisherApex 両システムで抽出したサンプルのlet7eおよびmir-16アッセイのCt値の比較データです。スタンダードプロトコルおよび大容量プロトコルで抽出したいずれにおいても、Ct値の差はすべて1サイクル範囲内にあることを示しています。
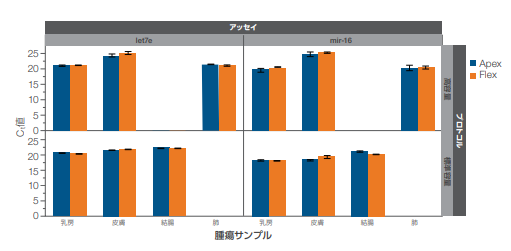
図7. 大容量プロトコル(上)およびスタンダードプロトコル(下)で抽出したサンプルの miRNA 標的の qPCR 結果。
KingFisher FlexおよびKingFisher Apexの両システムを用い、スタンダードプロトコルで乳房、皮膚および結腸から、大容量プロトコルで乳房、皮膚および肺のそれぞれの FFPE サンプルを用いてmiRNA の抽出を行いました。いずれのプロトコルにおいても、TaqMan microRNA RTキットを使用してcDNAテンプレートを合成し、let7eおよびmir-16を標的としたqPCRを実行してサンプル中のmiRNAを解析しました。
MagMAX Multi-Sample Ultra 2.0 Isolation Kitを用いたDNA抽出結果の比較
KingFisher ApexおよびKingFisher FlexシステムでMagMAX Multi-Sample Ultra 2.0 Isolation Kit を使用して、全血、バフィーコートおよび口腔粘膜スワブからの DNA の抽出を行いました。全血では、2 検体のサンプルを使用し、サンプル容量を振り(200 µL、400 µL、1 mLおよび 2 mL)、各容量に最適化されたプロトコルでの抽出を行い、DNA 収量および純度比を求めました。バフィーコートは 3 検体のサンプルを使用し、バフィーコート用のプロトコルで抽出を行いました。抽出したサンプルはQubit 4.0 Fluorometerを使用してDNA収量を測定し、NanoDrop 8000微量分光光度計を使用して純度比をそれぞれ求めました。口腔粘膜スワブは6 検体のサンプルを使用し、口腔粘膜スワブ用のプロトコルで抽出を行いました(1回の抽出に1スワブを使用)。抽出したサンプルはQubit 4.0 Fluorometerを使用してDNA収量を測定し、NanoDrop 8000微量分光光度計を使用して純度比を測定しました。図8は、全てのプロトコル(さまざまなインプット容量の全血、バフィーコートおよび口腔粘膜スワブ)における収量の詳細を示しています。全サンプルともにDNA 収量はKingFisher FlexおよびKingFisher Apexの両システム間で同等でした。
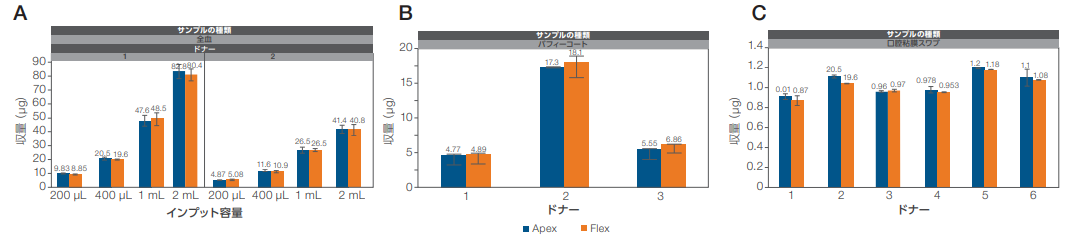
図8. KingFisher FlexおよびKingFisher ApexシステムでMagMAX Multi-Sample Ultra 2.0 Kitを使用して抽出したサンプルのDNA収量
(A)さまざまなインプット量の全血、(B)バフィーコート(C)口腔粘膜スワブ。
NanoDrop dsDNAモジュールを使用して、全抽出サンプルの純度の解析を行いました。KingFisher FlexおよびKingFisherApex の両システムを用いて処理した全血サンプル(200 µL、400 µL、1 mLおよび2 mLのインプット容量)、バフィーコートサンプルおよび口腔粘膜スワブサンプルの抽出物は、それぞれ同等の吸光度比(A260/A280およびA260/A230)を示しました(図9および10)。この結果は、KingFisher Apexシステムで処理したサンプルとKingFisher Flexシステムで処理したサンプルのDNA の純度が同等であることを示しています。図10は、口腔粘膜スワブ由来の抽出サンプルのA260/A230が全体的に低いことを示しています。KingFisher FlexおよびKingFisher Apex両システムから得られたデータがサンプル間で適切な標準偏差範囲にあることから、この吸光度比の低さは装置プラットフォームに起因するものではなく、サンプル間のばらつきに起因するものであることを示しています。
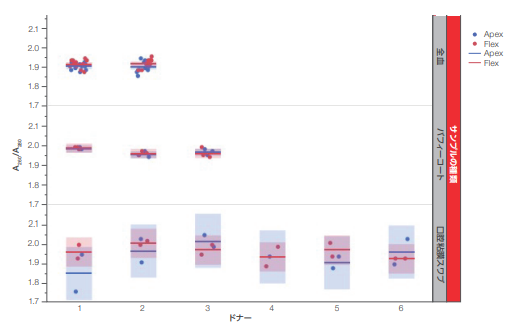
図 9. NanoDrop 8000 微量分光光度計で測定した各サンプルの純度。
全血サンプル(200 µL、400 µL、1 mLおよび2 mLのインプット容量)、バフィーコート、口腔粘膜スワブから抽出した全サンプルのA260/A280値。
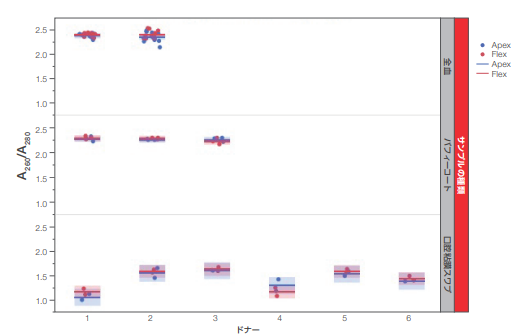
図10. NanoDrop 8000微量分光光度計で測定した各サンプルの純度。
全血サンプル(200 µL、400 µL、1 mLおよび2 mLのインプット容量)、バフィーコート、口腔粘膜スワブから抽出した全サンプルのA260/A230値。
MagMAX mirVana Total RNA Isolation Kitを用いたRNA抽出結果の比較
KingFisher ApexおよびKingFisher FlexシステムでMagMAX mirVana Total RNA Isolation Kitの細胞・組織プロトコルを使用して、2.5×105、5×105、106個のHeLa細胞およびHuh7細胞からRNAの抽出を行いました。同様に、2検体の全血サンプルから生体液プロトコルを使用して抽出を行いました。抽出したRNAサンプルは、Qubit RNA HS Assay Kitを使用してRNA収量を測定し、qPCRを実行してmRNAおよびmiRNA標的の検出を行いました。さらにAgilentバイオアナライザを用いてRIN(RNA integrity number)値およびピークプロファイルを解析し、RNAの完全性およびサイズを確認しました。図11は、HeLa細胞およびHuh7細胞から抽出したRNAサンプルと全血から抽出したRNAサンプルを Qubit RNA HS Assay Kitで測定したトータル RNA の収量を示します。全ての各細胞と細胞密度において、KingFisher Apexシステムを使用して抽出したサンプルの収量は、KingFisher Flexシステムを使用して抽出したサンプルの同等以上でした。
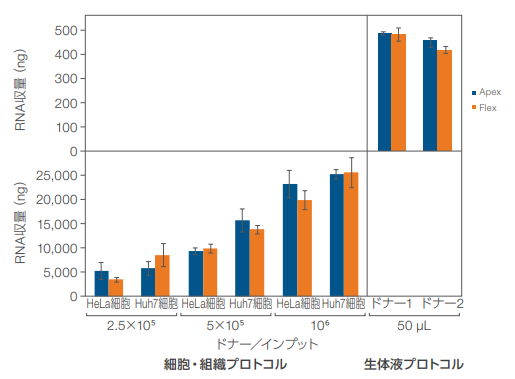
図 11. 細胞・組織プロトコルと生体液プロトコルを使用して抽出したサンプルのトータルRNAの収量(ng)。
MagMAX mirVana Total RNA Isolation Kitを使用して、2.5×105、5×105、106個のHeLa細胞およびHuh7細胞について細胞・組織プロトコルを用いてRNAを抽出し、2検体の全血サンプル(各50 µL)は生体液プロトコルを用いてRNAを抽出しました。
qPCRの解析には、Invitrogen ™ SuperScript ™ VILO ™ Master Mix を使用して、HeLa 細胞、Huh7 細胞、全血から抽出したRNA サンプルの逆転写(RT)反応を行いました。RT 反応後、Applied Biosystems™ TaqMan® Universal Master Mix II, with no UNGを用い、βアクチン(m1_ACTB)およびGAPDH(m1_GAPDH)を標的としたmRNA 測定用 TaqMan®プライマーを使用して qPCR 反応を行いました。図 12 は、2.5×105、5×105、106個の HeLa 細胞および Huh7 細胞由来の RNAサンプル、全血由来のRNAサンプルを用いたqPCRのCt値をまとめた結果ですが、いずれのmRNA標的アッセイにおいてもシステムプラットフォーム間でCt値の差は1サイクル以内となっており、同等な結果が得られています。また、図 12では、ACTB 標的としたアッセイではHeLa細胞およびHuh7細胞のインプットが増加しても、予想される増幅の早期化やCt値の低下が確認できませんでした。これはKingFisher FlexとKingFisher Apexのいずれのシステムにおいても観察されていることから、装置由来の問題ではないと考えられます。
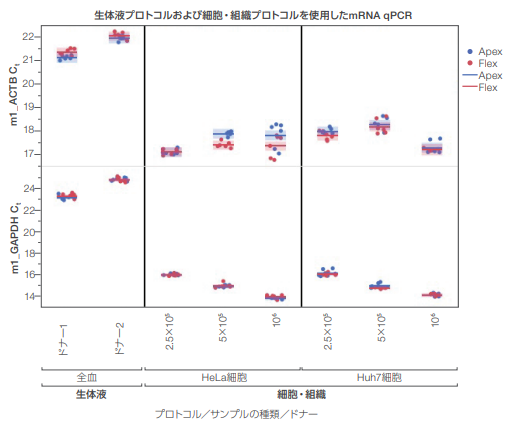
図12. 細胞・組織プロトコルおよび生体液プロトコルを使用して抽出した核酸の mRNA標的(m1_ACTBおよびm1_GAPDH)の qPCR。
2.5×105、5×105、106個の HeLa 細胞および Huh7 細胞と、2 検体の全血サンプル(各 50 µL)から、KingFisher Apex および KingFisher Flexシステムを使用して抽出したRNA サンプルを用い、mRNA 標的(m1_ACTBおよびm1_GAPDH)のqPCR解析を行いました。
miRNA の検出テストでは、専用の TaqManプライマーと試薬を使用してRT 反応を行い、miRNA 標的であるmir-24とmir16を検出しました。KingFisher ApexおよびKingFisher FlexシステムでMagMAX mirVana Total RNA Isolation Kitを使用してHeLa細胞、Huh7細胞および全血から抽出したRNAサンプル中の mir-24 および mir-16を標的に検出するために、逆転写反応後、TaqMan Universal Master Mix II, with no UNGによるqPCR反応を行いました。図13は、mir-16とmir-24を標的としたmiRNA qPCR の Ct値が KingFisher Apex およびKingFisher Flexシステム間で同等であることを示しています。さらにKingFisher Apexシステムで抽出したRNAサンプルは、KingFisher Flexシステムで抽出したRNAサンプルと比較して、インプットサンプル量の多いサンプル(106細胞)で、HeLa 細胞およびHuh7細胞ともに、どちらのmiRNA標的でもより低いCt値を示しました。
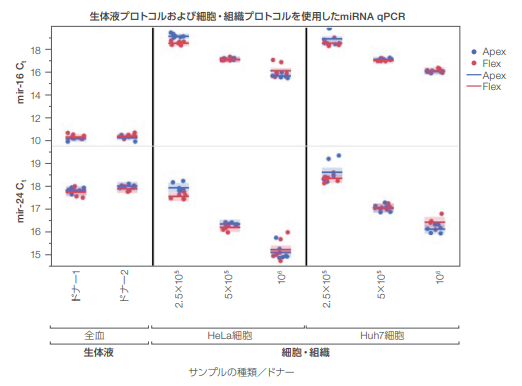
図13. 細胞・組織プロトコルおよび生体液プロトコルを使用して抽出した核酸のmiRNA標的(mir-16およびmir-24)のqPCR。
miRNA標的のqPCRでは、KingFisher ApexおよびKingFisher Flexシステムを用い、2.5×105、5×105、106個のHeLa細胞およびHuh7細胞ならびに2 検体の全血サンプル(各 50 µL)から抽出したRNAサンプルを用い、mir-16およびmir-24を標的としたアッセイを行いました。
AgilentバイオアナライザとAgilent™ RNA 6000 Nanoキットを使用して、HeLa細胞およびHuh7細胞から抽出したRNAサンプルのピークプロファイルとRIN値を評価しました。図14は、KingFisher ApexおよびKingFisher Flexシステムで抽出したトータルRNAが同等のサイズプロファイルのRNAを回収できていることを示しています。RIN 値も同等であることを示しています。
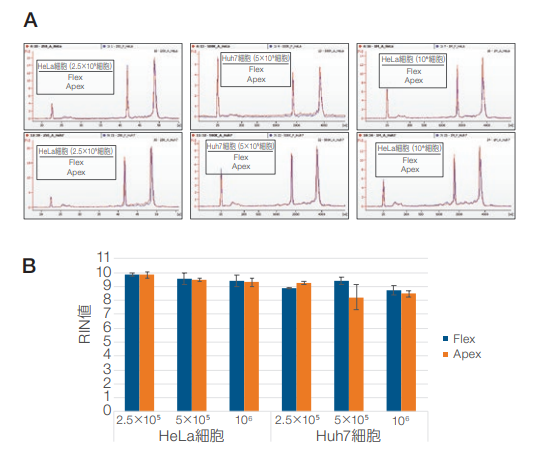
図 14. Agilentバイオアナライザを用いたRNA のサイズおよび完全性の確認。
(A)さまざまなインプット量のHeLa細胞およびHuh7細胞から抽出したRNAサンプルを、RNA 6000 Nano KitとAgilentバイオアナライザを用いて解析しました。(B)RIN値を測定し、サンプルの完全性を評価しました。
MagMAX Cell-Free DNA Isolation Kitを用いたcfDNA抽出結果の比較
KingFisher ApexおよびKingFisher Flexシステムを使用して、MagMAX Cell-Free DNA Isolation Kit用の2 mLおよび4 mLプロトコルを用いて、3 検体のドナー由来の血漿サンプルからcfDNAを抽出しました。図 15 は、KingFisher Flex およびKingFisher Apexシステム間のcfDNAの収量とサイズプロファイルの詳細を示しています。フラグメントのサイズは、AgilentバイオアナライザとAgilent ™ HS DNA Assayを使用して評価しました。KingFisher Apexシステムで抽出したcfDNAサンプルでは、コンタミネーションしたgDNAに対するcfDNAの割合が、KingFisher Flexシステムを使用した場合の同等以上であることが明らかになりました。
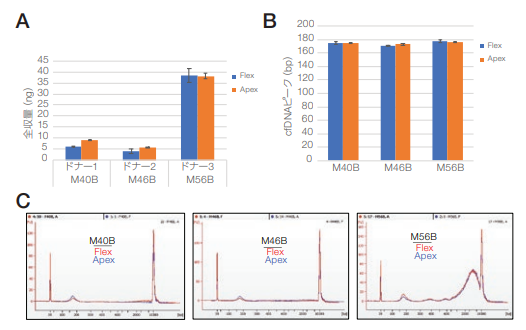
図15. KingFisher FlexおよびKingFisher Apexシステムでの3検体のサンプルから抽出したcfDNAの収量およびサイズの比較(2 mLプロトコルを使用)。
(A)3検体のサンプル由来のcfDNA収量はQubit dsDNA HS Assayを使用して解析しました。cfDNAの(B)ピークサイズ(bp)および(C)プロファイルはAgilent HS DNA Assayを使用して解析しました。
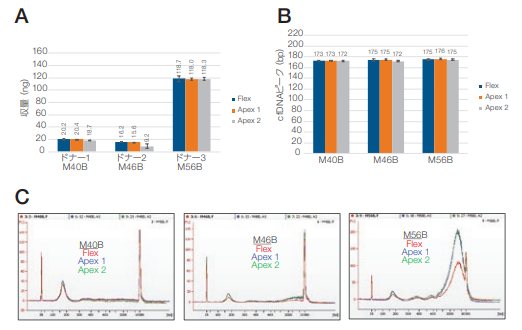
図16. KingFisher FlexおよびKingFisher Apex(2台使用)システム間の、3検体のサンプルから抽出したcfDNAの収量およびサイズの比較(4 mLプロトコルを使用)。
(A)3 検体のサンプル由来の cfDNA収量は Qubit dsDNA HS Assayを使用して解析しました。cfDNAの(B)ピークサイズ(bp)および(C)プロファイルはAgilent HS DNA Assayを使用して解析しました。
2 台の KingFisher Apexシステムで、同時に4 mLプロトコルの抽出を行うことでシステム間の違いを確認しました。Qubit HS dsDNA AssayおよびAgilent HS DNA Assayを使用してcfDNA の収量およびサイズを解析しました。図 16で、3 台のシステム(KingFisher Flexシステム1台とKingFisher Apexシステム2 台)の間の同等な結果が得られましたが、KingFisher Apexシステム2台のうちの1台(「Apex 2」)で、1検体のサンプルで収量が低く、ばらつきが大きくなっています。これは血漿の品質に関連している可能性が高く、装置のパフォーマンスには関連していないと考えられます(図16)。
MagMAX Cell-Free Total Nucleic Acid IsolationKitを用いた核酸抽出結果の比較
KingFisher ApexおよびKingFisher Flexシステムを使用して、MagMAX Cell-free Total Nucleic Acid Isolation Kit用の2 mLおよび4 mLプロトコルを用いて、血漿由来のセルフリー核酸(DNA/RNA)を抽出しました。図 17は、2 mL プロトコルを用いて抽出した核酸サンプルについてHS DNA AssayとAgilentバイオアナライザを使用して cfDNAピークサイズの解析を行った結果です。KingFisher FlexおよびKingFisher Apexシステムで収量およびサイズが同等であることを示しています。2つのプラットフォーム間のcfDNAの割合も同等であることが確認できました。1 検体の F58C 由来の抽出サンプルでは、両システムともに上側のマーカーと重複する大きなgDNAピークが存在し、cfDNAピークの正確な定量が妨げられたため、cfDNA率の解析から除外しました。
4 mLプロトコルを用いて抽出した核酸サンプルについても、Qubit dsDNA HS AssayとAgilentバイオアナライザを使用して、DNA のピークサイズとcfDNA 率の解析を行いました。図18は、各ドナーサンプルにおいて、KingFisher Apexシステムで抽出したサンプルをQubit dsDNA HS Assayで測定した結果、トータルのcfDNA収量が、KingFisher Flexシステムで抽出したサンプルの結果と同等であることを示しています。サンプルM56BおよびF58Cでは、gDNA含有量が多いためにばらつきと標準偏差が大きく、Agilentバイオアナライザ上で上側マーカーが不明確になり、cfDNA率の算出が不可能でした。Agilentバイオアナライザで算出したcfDNAピークは、KingFisher FlexおよびKingFisher Apexシステムで同等でした。
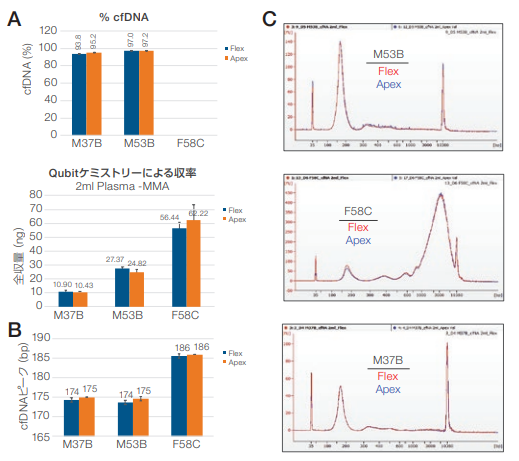
図17. KingFisher FlexおよびKingFisher Apexシステムを使用した、2 mLプロトコルでのcfDNAの収量およびサイズの比較。
(A)3検体のサンプルのcfDNA収量を、Qubit dsDNA HS Assayを使用して解析しました。cfDNA の(B)ピークサイズおよび(C)プロファイルをAgilent HS DNA Assayを用いて解析しました。サンプル F58CではgDNA含有量が高いために上側マーカーが不明確となり、cfDNAの定量が不可能でした。これは、KingFisher ApexおよびKingFisher Flexの両システムで共通して生じていることから、サンプルの品質に起因するもので、装置に起因するものではないと考えられます。
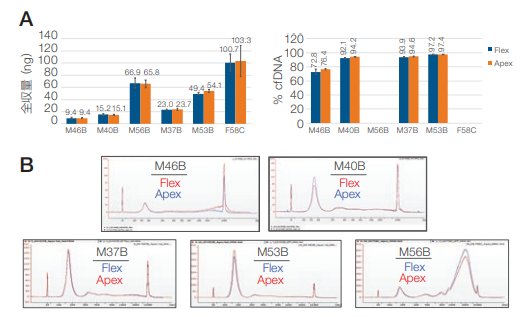
図18. KingFisher FlexおよびKingFisher Apexシステムを使用した、4 mLプロトコルでのcfDNAの収量およびサイズの比較。
(A)6検体のサンプルのcfDNA収量を、Qubit dsDNA HS Assayを使用して解析しました。サンプルM56BおよびF58CではgDNA含有量が多いために上側マーカーが不明確となり、cfDNAの定量が不可能でした。これは、KingFisher ApexおよびKingFisher Flexの両システムで共通して生じていることから、サンプルの品質に起因するもので、装置に起因するものではないと考えられます。(B)Agilent HS DNA Assayを使用して、cfDNAのピークサイズ(bp)とプロファイルを解析しました。
トータルcfRNAは、QuantStudio 12 K Flexシステムを用い、m1_GAPDHおよび m1_ACTBを標的とするTaqMan Assayを使用したqPCRによって解析しました。図 19は、m1_ACTBおよび m1_GAPDH 標的を使用した、2 mLおよび 4 mL 抽出プロトコルによるデータを示します。2 mLプロトコルで抽出したサンプルでは、ACTBおよびGAPDHのいずれの標的に関しても、KingFisher Flexおよび KingFisher Apexシステムで同等の結果が得られました。4 mLプロトコルで抽出したサンプルでは、ドナーサンプルM40Bを除き、KingFisher FlexおよびKingFisher Apexシステム間で同等な結果が得られました。ドナーサンプルM40BのCt値は、KingFisher Apexシステムによる抽出でより高く(増幅が遅く)、標準偏差の範囲を超えていました。そこで、3 検体のサンプルを追加して2 回目の抽出を行いました。追加した3つのサンプルに関しては、KingFisher FlexおよびKingFisher Apexシステムで、ACTBおよびGAPDHのいずれの標的においてもCt値は同等でした。したがって、1回目の抽出結果のばらつきは操作者またはサンプルのエラーに起因し、装置に起因するものではないことが示唆されます。
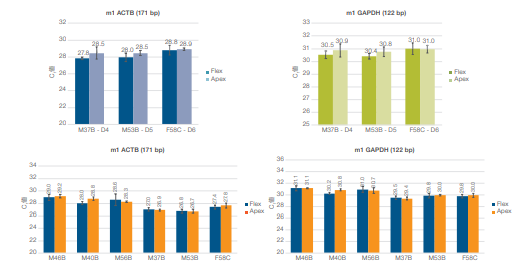
図19. 血漿cfサンプルから2 mLおよび4 mLプロトコルを使用して抽出した cf 核酸(DNA / RNA)を用いた、GAPDH および ACTBを標的としたqPCR。
MagMAX Cell-Free Total Nucleic Acid IsolationKitを使用して2 mLおよび4 mLプロトコルを使用してcf核酸(DNA /RNA)を抽出し、m1_ACTBおよびm1_GAPDHを標的として解析を行いました。
ストレージチューブへの溶出効率の比較
溶出効率のテストは、MagMAX DNA Multi-Sample Ultra 2.0Kit 用の 2 mL プロトコルを用い、全血からの DNA 精製で行いました。この実験では、KingFisher Apexシステムを用いたストレージチューブへの溶出、KingFisher FlexシステムおよびKingFisher Apexシステムを用いた24ディープウェルプレートへの溶出について設定し、溶出効率の比較を行いました。図20 では、同一ドナーサンプルから抽出した各 DNA サンプルのdsDNAおよびssDNAの品質の評価として、Qubit HS DNAAssayおよびNanoDrop 8000微量分光光度計で測定した収量および純度の結果をまとめています。KingFisher Apexシステムでのストレージチューブへの溶出効率とKingFisher Flexシステムでの溶出効率に若干差がみられますが、これはストレージチューブへの溶出では24ディープウェルプレートと比較してサンプル温度が低く抑えられているためと考えられます。A260/A280およびA260/A230の吸光度比とDNAサイズは、3つのプラットフォーム間で同等であることが示されています。これは、バイオバンキングや核酸の長期保存に有用なストレージチューブ(ST)への溶出のパフォーマンスが、ディープウェルプレートへの溶出と同等であることを示しています。
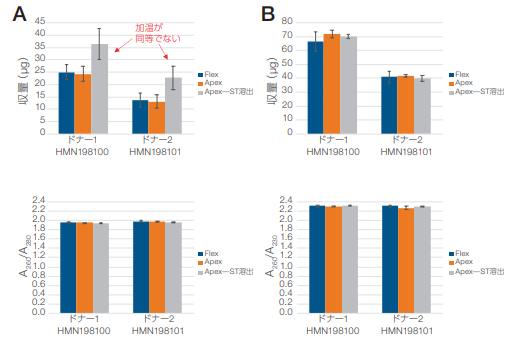
図20. MagMAX DNA Multi-Sample Ultra 2.0を使用した2 mLプロトコルでの全血サンプルの抽出における、ストレージチューブ(ST)への溶出およびディープウェルプレートへの溶出間の、DNA の収量および純度の比較。
KingFisher FlexおよびKingFisher Apexシステムで24ディープウェルプレートへの溶出およびKingFisher Flexシステムでストレージチューブへ溶出させたサンプルに対して、(A)Qubit HS DNA AssayでdsDNAの収量、(B)NanoDrop 8000微量分光光度計でssDNAの収量(C)A260/A280および(D)A260/A230を測定しました。
冷却機能の性能確認
KingFisher Flexシステムに搭載されている効率的な加温機能に加えて、KingFisher Apexシステムでは新たに冷却ブロックが追加されています。冷却機能は抽出後のサンプル品質の維持およびタンパク質抽出に有用です。この冷却機能の性能を確認するために、水500 µLを添加した24ディープウェルプレートの角の2つのウェルおよび中央の2つのウェルに、ColeParmer™ Digi-Sense™ Thermocouple温度計とFisherbrand™ Type-K Digital Thermometer Probeを設置し、16時間後に温度プロファイルを読み取りました。また 96 ディープウェルプレートでも同様の試験を行うため、水 100 µLを添加して同等の試験を行いました(図21)。
16 時間後、KingFisher Apexシステムに設置したDigi-SenseThermometerの解析を行いました。図22は、全てのサンプルが設定した時間内に8℃を下回る温度に到達していることを示しています。16時間後、冷却ブロック内に結露が観察されました。この結露は、水平を保った装置の冷却ブロックの片側に局在していました。このためインキュベーション時間が長い場合には、冷却温度は4℃より高い温度に設定する必要があります。
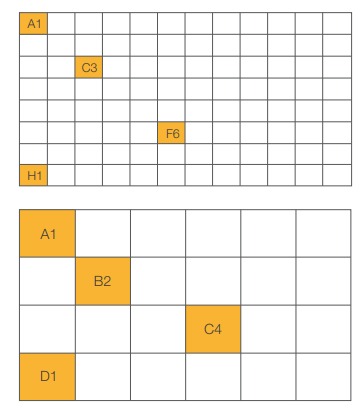
図21. KingFisher Apexシステムの冷却および加温プロファイルを確認するための96ディープウェルプレートおよび24ディープウェルプレートに設置した熱電対プローブのポジション。
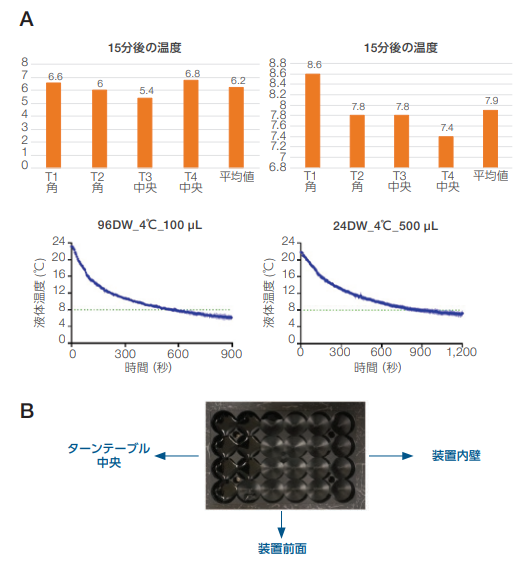
図22. 経時的に測定した冷却プロファイルと結露の発生。
(A)KingFisher Apexシステムで4℃に設定した96ディープウェルプレートおよび24ディープウェルプレート中の100 µLおよび500 µLの水の冷却プロファイル
(B)16時間後の冷却ブロック内の結露の状況
UVランプによる汚染除去
KingFisher Apex システムには汚染除去アプリケーションのニーズに対応した UV ランプが新たに搭載されています。KingFisher Apexシステムの 30 分間の汚染除去プロトコルを実行し、ATCC から入手した GFP 発現 E. coli を使用してKingFisher Apexシステムの汚染除去効率を評価しました。また比較対象としてGrant Instruments™ Grant BIO™ DNA/RNA UVC/T-M-ARキャビネットを使用しました。この試験では、0.1 mg/mLアンピシリンを含むLB培地 5 mLに大腸菌を播種し、200 rpmで振とうしながら30℃で一晩インキュベーションしました。約 16 時間後、培養液から1 mLを採取して1:10に希釈し、この希釈サンプル200 µLを96ウェルプレートに移し、Thermo Scientific™ Multiskan™ SkyHigh吸光マイクロプレートリーダーを使用して600 nmで測定し、大腸菌の光学密度を求めました。
一晩培養した培養液を用いて、OD600が約 0.1になるように調製しE. coli GFP 培養を開始しました。OD600が0.58に達するまで、150 rpmで振とうしながら37℃でインキュベーションしました。10-7までの 10 倍希釈系列サンプルを調製し、各試験(Apex UV、Grant BIO UVおよびUV非照射)用のプレートに添加しました。蓋を外した10枚のプレートをKingFisher Apexシステム内に(図23)、同濃度の10枚のプレートをGrant BIOキャビネット内にそれぞれ設置し、30分間UVランプを照射しました。さらに、UVランプに曝露させない5枚のコントロールプレートをシステム外に放置しました。30分間のUVランプへの曝露後、LBプレートに蓋をして37℃で一晩インキュベーションしました。16 時間後、コロニーを計数しました。KingFisher Apexシステム内に設置したプレート上にもGrant BIOキャビネットに設置したプレート上にもコロニーは認められませんでした。一方、システム外に放置したプレートには1プレートあたり約200コロニー、平均2.1×109 CFU/mLに相当する顕著な増殖が認められました。

図 23. KingFisher Apexシステム内の汚染除去試験におけるLBプレート配置の例。
結論
本稿では、核酸抽出において KingFisher Apex システムとKingFisher Flexシステムが同等な性能であることを確認できました。テストでは、磁気ビーズベースのMagMAX DNA MultiSample Ultra 2.0 Kit、MagMAX Cell-Free DNA Isolation Kit、MagMAX Cell-Free Total Nucleic Acid Isolation Kit、MagMAX mirVana Total RNA Isolation KitおよびMagMAX FFPE DNA/RNA Ultra Kitを用いてさまざまなタイプのサンプルから核酸を抽出しました。回収された核酸サンプルの質、収量および純度は、Qubit Fluorometer、Agilentバイオアナライザシステム、NanoDrop 8000微量分光光度計を用いて解析しました。またRT-qPCRおよび qPCR の解析においても、2つのシステム間でDNA、mRNAおよびmiRNAの検出レベルが同等であることが示されました。KingFisher Apexシステムでは、バイオバンキング用ストレージチューブへ直接溶出ができます。溶出した核酸サンプルの収量および純度の比較では、KingFisher ApexシステムとKingFisher Flexシステムで差が見られなかったことから、KingFisher Apexシステムは、ストレージチューブおよびプレートいずれのフォーマットへの溶出にも適していることが示唆されます。また新たに搭載された冷却機能により、保管前の溶出プレートを冷却状態で置くことができるため、抽出後の核酸の品質をより確実に保持することができます。しかしながら長時間にわたる冷却では4℃より高い温度に設定する必要があります。KingFisher Apexシステム内のUVランプに30分間曝露したプレート上でコロニー増殖が認められなかったことから、本システムの滅菌機能の有効性も確認できました。高タイターのサンプルからの抽出処理では、装置内に設置されたUVランプによる滅菌は極めて有用です。