ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- HOME
- ライフテクノロジーズジャパン株式会社 バイオサイエンス事業部
- 製品・サービス詳細
- KingFisher PlasmidPro Maxi Processorを使用して簡単にMaxiスケールでのプラスミド精製を実現
会社カテゴリー:研究関連資材
主サービス提供地域:
製品・サービス詳細
KingFisher PlasmidPro Maxi Processorを使用して簡単にMaxiスケールでのプラスミド精製を実現
サービスカテゴリー:分析、研究・開発、モダリティ
KingFisher PlasmidPro Maxi Processorを使用して簡単にMaxiスケールでのプラスミド精製を実現
• 遠心分離不要:プラスミドDNA(pDNA)の精製に遠心分離や吸引ろ過は必要ありません。
• 革新的な技術:pDNAの精製に必要な試薬がセット済みのオールインワンカートリッジを使用し、
5分以内に装置へのセットアップが可能です。
• 完全自動化:培養液を注入し、Startボタンを押すだけで、pDNAを75分以内に精製できます。
• エンドトキシンフリー:エンドトキシンレベルが <0.1 EU/μgのpDNAを精製します。
はじめに
プラスミドDNAの精製は、分子生物学研究において重要な役割を担っています(図1)。 例えば、Maxiスケールで単離されたpDNAは、mRNA合成、ワクチン開発、抗体生産、細胞治療、遺伝子治療やウイルスベクター生産など、さまざまな分野で用いられています( 図 2)。このような分子生物学的な手法で利用するための高品質なpDNAの需要が高まるにつれ、信頼性の高いpDNA精製の自動化プロセスの重要性が増しています。pDNAは、従来のカラムベースの方法で効率的に精製することができます。しかし、カラムを用いた精製は、遠心分離や真空ろ過を何度も行う必要があるため、pDNAを単離するためには多くの労力と時間がかかり、研究者にとってボトルネックとなっています。
Thermo Scientific™ KingFisher™ PlasmidPro Maxi Processor は、pDNA精製のボトルネックとなっている部分を完全自動化で減らすことで、精製ワークフローを合理化できます(図1)。 KingFisher PlasmidPro Maxi Processorは、Invitrogen™ PureLink™ Expi Endotoxin-Free Maxi Plasmid Purification Kitと同じ試薬が充填済みのオールインワンカートリッジを使用する当社最新のプラスミド精製システムです。革新的なテクノロジーとユーザーフレンドリーなインターフェースを備えたこのシステムは、効率的で信頼性の高いMaxiスケールのpDNA精製を実行するための強力なツールです。シンプルなワークフローにより貴重な時間を節約し、要求の厳しいダウンストリームアプリケーション用の高品質pDNAを得るために必要なリソースも少なくて済みます。ここでは、2.7 kbから11.4 kbまでの4種類のプラスミドをMaxiスケールで精製するKingFisher PlasmidPro Maxi Processorの性能について包括的に説明します。性能については、pDNAの収量や純度、スーパーコイル状pDNAの品質、トランスフェクション効率の点で評価しています。
• カラムを用いた手動でのプラスミド精製のワークフロー
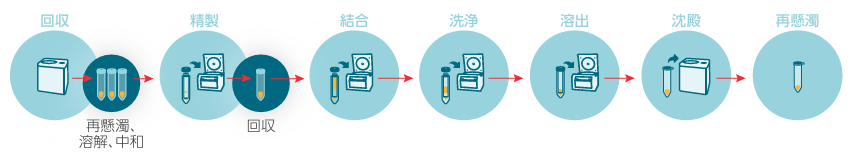
• KingFisher PlasmidPro Maxi Processorを用いた全自動のプラスミド精製のワークフロー
図1. 手動と全自動のプラスミド精製のワークフローの比較。
カラムベースのプラスミド精製は効果的なワークフローですが、多くの手作業が必要となり、時間と労力がかかるのに対して、KingFisher PlasmidPro Maxi Processorは使い方が簡単で、75分以内に全自動でプラスミド精製が可能。
材料および方法
バクテリアの培養
Invitrogen™ One Shot™ TOP10 Chemically Competent E.coli を用いてプラスミドA(~2.7 kb)、プラスミドB(~5.1 kb)、またはプラスミドD(~11.4 kb)を 、Invitrogen™ MAX Efficiency™ DH5α Competent Cells(大腸菌)を用いてプラスミドC (~6.6 kb)を形質転換しました。形質転換後に得られた各プラスミドの単一のコロニーを一晩培養し、グリセロールストックを調製しました。グリセロールストックは-80 ℃で保存し、pDNA単離のための細菌培養の準備に用いました。培養はGibco™ LB Broth(1X)を用い、37 ℃のインキュベーター内で225 rpmで連続的に振とうしながら一晩培養しました。培地は、プラスミドBで形質転換された細胞を選択するために100 μg/mLカルベ ニシリン、プ ラスミド AまたはプラスミドCで形質転換された細胞を選択するために100 μg/mLアンピシリン、プラスミドDで形質転換された細胞を選択するために50μg/mLカナマイシンを添加しました。培養物の光学濃度は、14~16時間の増殖後、Thermo Scientific™ NanoDrop分光光度計で測定しました。
pDNAの分離
pDNA は、Thermo Scientific™ KingFisher™ PlasmidPro Maxi Processor Endotoxin-Free CartridgeとKingFisher PlasmidPro Maxi Processorを用いて精製しました。プラスミドA、B、C、Dの各培養液を一晩培養し、それぞれ150 mLを処理しました。
異なる9台のKingFisher PlasmidPro Maxi Processor(プラスミドA:n=19、プラスミドB:n=13、プラスミドC:n=21、プラスミドD:n=21)で 行 っ た 74の抽出を用いてデータを分析しました。このシステムを用いて精製したpDNAの品質は、同じ培養液からカラムベースのPureLink Expi Endotoxin-Free Maxi Plasmid Purification Kitを用いて手動で単離したpDNAと比較しました。
pDNA分析
溶出したサンプルをThermo Scientific™ NanoDrop™ Eight微量分光光度計を用いて分析し、pDNA収量とA260 /A280、A260 / A230を測定しました。各プラスミドのサブセットを1% アガロースゲルを用いて75 Vの定電圧で75分間泳動し、サンプル中のスーパーコイル状pDNA、ゲノム DNA、RNAの混入量を視覚的に評価しました。スーパーコイル状pDNAは、Invitrogen™ iBright™ FL1500 Imaging Systemで取得した画像を用いた濃度測定により定量し、JMP™ ソフトウエア16.0を用いてデータを解析しました。
エンドトキシン濃度測定
Endosafe™ nexgen-PTS™ endotoxin testing systemを用いて、4種類のプラスミドを網羅した代表的な26サンプルをエンドトキシンの存在について分析しました。
結果は、手動カラムベースのプラスミド精製プロセスで得られたpDNAサンプルのエンドトキシンレベルと比較しました。
精製pDNA中の大腸菌由来残存ゲノムDNA(gDNA)
各サンプルに残存する大腸菌gDNAを測定し、プラスミド調製物の純度を評価しました。分析には、大腸菌ゲノムのシングルコピー遺伝子をターゲットとするApplied Biosystems™ TaqMan™ Ba04646242_s1 ECs4157 qPCRアッセイと Applied Biosystems™ TaqMan™ Fast Advanced Master Mixを使用しました。
大腸菌のコントロールであるTOP10株またはDH5α株のgDNAを連続希釈し、濃度に対するCt値の標準曲線を作成し、プラスミド調製物中のgDNAの割合を推定しました。精製したpDNAサンプルを100倍希釈し、サンプルから2 μLずつ3連の反応で試験しました。データはJMPソフトウエア16.0を用いて解析しました。
pDNAトランスフェクション効率
KingFisher PlasmidPro Maxi Processorで精製したpDNAのトランスフェクションの効率を、手作業で精製した場合と比較しました。低グルコースおよび10% FBSを含むDMEMで維持したHuH-7細胞へのトランスフェクションは、Invitrogen™ Lipofectamine™ 3000 Transfection ReagentおよびGibco™ Opti-MEM™ ReducedSerum Mediumで希釈した100 ngの精製したプラスミドBまたはCを用いて、1ウェルあたり10,000細 胞( n=6)で行いました。プレートは37 ℃、5% CO2で20~24時間インキュベートしました。
分泌型アルカリホスファターゼ(SEAP)レポーター配列を含むプラスミドBについて、培養液25 μLを標準的なプロトコルに従い、Invitrogen™ Phospha-Light™ SEAP Reporter Gene Assay Systemを用いて遺伝子発現を測定しました。
アッセイで生じた発光は、FLUOstar™ Omegaマイクロプレートリーダーを用いて測定しました。Invitrogen™ Luc Screen™ Extended-Glow Luciferase Reporter Gene Assay Systemを用いて、ルシフェラーゼ配列を含むプラスミドCのトランスフェクション効率を評価しました。ルシフェラーゼの発光は、細胞を室温で10分間インキュベートした後、培養プレート中で測定しました。
データはJMPソフトウエア16.0を用いて解析しました。
結果
プラスミドA、B、C、Dで形質転換した培養物を14~16時間培養した後の光学密度(OD600)は、それぞれ2.3、2.8、3.1、2.8でした。KingFisher PlasmidPro Maxi Processorでの精製後に得られた全pDNA収率を図2-Aに示します。培養液の量が多くても、自動精製プロセスで単離された全プラスミドAの総収量は、手動精製プロセスで予想されるものと同様でした。全ての精製サンプルの平均A260 /A280およびA260 /A230比はそれぞれ1.8以上と2.0以上であり、一般的な下流のアプリケーションで必要な純度の要件を満たす結果が示されました(図2-B)
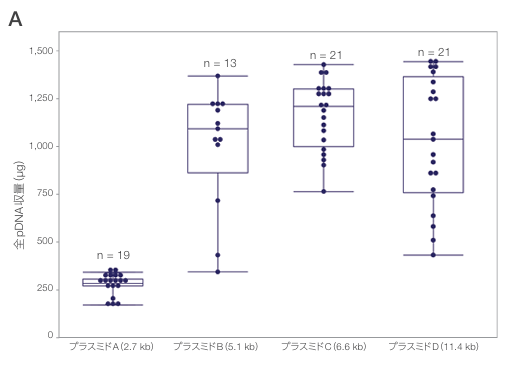
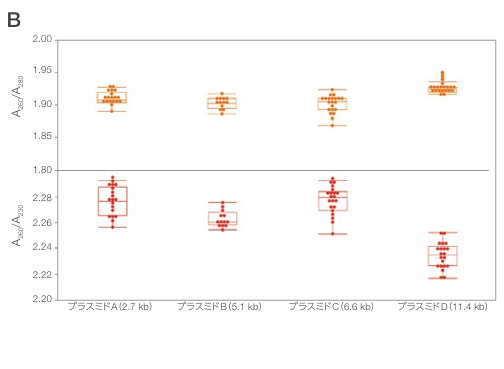
図2. KingFisher PlasmidPro Maxi Processorで精製した4種類のプラスミドの収量と純度。
(A)全pDNA収率。ボックスプロットはデータの分布を示し、各ドットは個々の精製pDNAを表す。(B)A260 /A280およびA260 /A230 pDNA純度比。
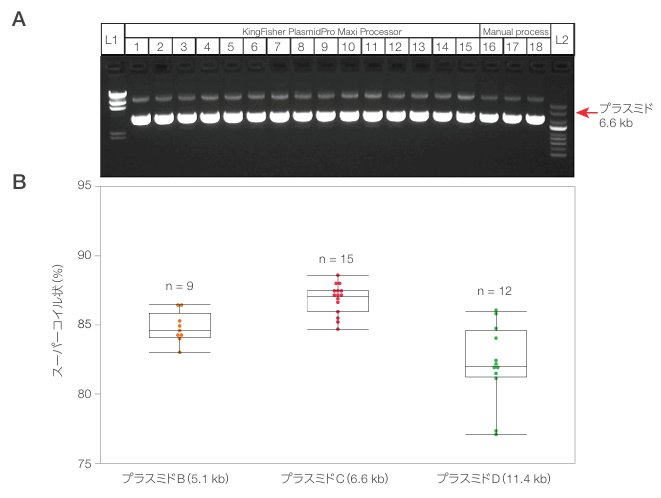
図3. pDNAアイソフォームのゲル電気泳動と濃度測定の結果。
(A)KingFisher PlasmidPro Maxi Processorを用いて精製したプラスミドC(6.6 kb、レ ー ン 1-15)のサンププラスミド6.6 kbルを、カラムベースのキットを用いて手動で精製したpDNA(レーン16-18)と 1%アガロースゲルで比較した。L1:直鎖化dsDNAラダー。L2:スーパーコイル状pDNAラダー。スーパーコイル状pDNAは6.6 kbで優勢だった。
(B)KingFisher PlasmidPro Maxi Processorを用いて精製したサンプルの一部におけるスーパーコイル状pDNAの割合。ボックスプロットはデータの分布を示し、各ドットは個々の精製を表す。3つのプラスミド全てについて、スーパーコイル状pDNAの平均割合は80%を超え、手動プロセスで精製したサンプルのスーパーコイル状pDNAの平均割合(88% )に匹敵した 。
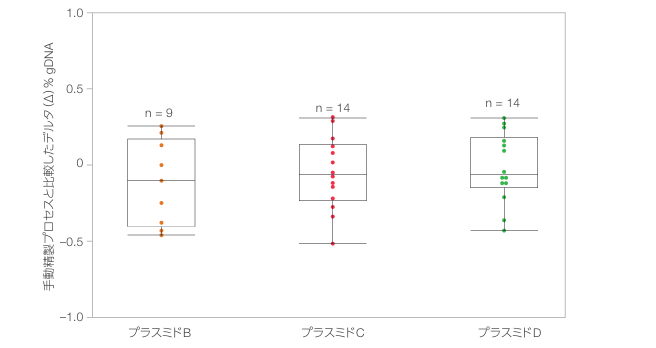
図4. 精製プラスミド調製物に残存する細菌gDNAの相対的な割合
KingFisher PlasmidPro Maxi Processorで精製したpDNAサンプルの残存gDNA量を示す代表的データ。各プラスミドの変化率は、同じプラスミドを手動で精製した後に残ったgDNA量に対する相対値である。ボックスプロットはデータの分布を示し、各ドットは個々の精製pDNAを表す。
残存大腸菌gDNA含有量
大腸菌gDNAは、プラスミド精製プロセス中に除去する必要があります。これは、残存する宿主細胞由来のDNAが下流のアプリケーションを妨害する可能性があるためです。そこで、qPCRでプラスミドB、C、お よび Dの37の代表的な精製サンプル中の残存大腸菌gDNA含量を測定しました。KingFisher PlasmidPro Maxi Processorで精製したサンプル中の残存gDNA量は、手動精製後に残った量の1%以内でした(図4)。このデータは、細菌由来のgDNAを除去するKingFisher PlasmidPro Maxi Processorの効率の良さを強く示しています。
エンドトキシン含有量
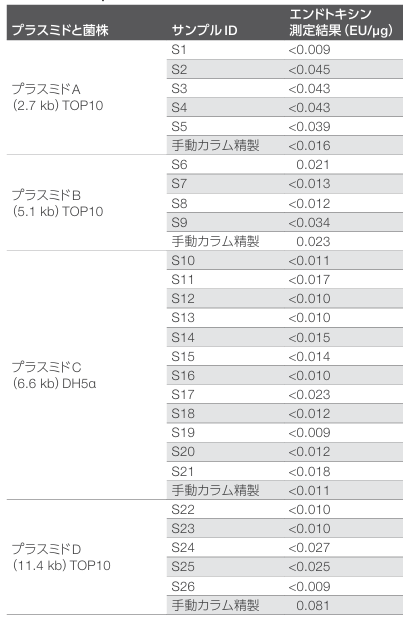
内毒素はグラム陰性菌の細胞壁に含まれるリポ多糖成分です。細胞溶解時に溶液中に放出され、精製によってpDNAと一緒に運 ば れ ま す( 図 4)。エンドトキシンレベルは細菌株、プラスミドタイプ、培養条件によって異なりますが、1 μgあたり0.1エンドトキシンユニット(EU)未満のpDNAはエンドトキシンを含まないトランスフェクショングレードとみなされており、創薬研究におけるバイオ医薬ワークフローに適しています。KingFisher PlasmidPro Maxi Processor用のカートリッジには、エンドトキシンを中和するエンドトキシン除去バッファーが含まれています。エンドトキシン含有量は、4つのプラスミドを全て含む26の代表的なpDNAサンプルで測定しました。サンプルS1~S26をKingFisher PlasmidPro Maxi Processorで精製し、サンプル中のエンドトキシンレベルをPureLink Expi EndotoxinFree Maxi Plasmid Purification Kitで手動精製した後の同じ培養からのサンプル中のエンドトキシンレベルと比較しました(表1)。 KingFisher PlasmidPro Maxi Processorで精製した全てのサンプルのエンドトキシンレベルは0.1 EU/µg未満となり、手動精製プロセスで分離したpDNAサンプルで測定したものと同等でした。
表1. KingFisher PlasmidPro Maxi Processorを使用して精製したpDNAサンプル中のエンドトキシン含有量
pDNAトランスフェクション効率
レポーター遺伝子を発現させるための精製pDNAの下流への応用は、遺伝子治療、機能ゲノミクス、タンパク質生産において重要です。KingFisher PlasmidPro Maxi Processorで精製したプラスミドBまたはプラスミドCをトランスフェクトした哺乳動物由来HuH-7細胞におけるレポーター遺伝子の発現を、手作業で精製したプラスミドBまたはプラスミドCをトランスフェクトした HuH-7細胞における同じ遺伝子の発現と比較しました。プラスミドBからのSEAPレポーター遺伝子およびプラスミドCからのルシフェラーゼ遺伝子の発現を比較したところ、KingFisher PlasmidPro Maxi Processorを用いたpDNA精製後と手作業による精製の結果と同等でした(図5)。こ れ ら の 結果は、効果的なpDNAの導入とレポーター遺伝子の発現を示しており、KingFisher PlasmidPro Maxi Processorで精製したpDNAは安定性が高く、哺乳動物細胞へのトランスフェクションに適していることを表しています。
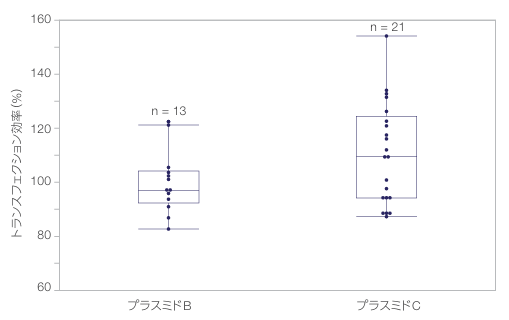
図5. 手動もしくはKingFisher PlasmidPro Maxi Processorで精製したプラスミドのトランスフェクション効率の比較。
プラスミドBもしくはCをHuH7哺乳類細胞のトランスフェクションに使用した。y軸の値は、手動精製プロセス(両プラスミドについて100%とした)を用いて単離したpDNAに対するトランスフェクション効率を示す。データは、KingFisher PlasmidPro Maxi Processorを用いて単離した100 ngのpDNAでトランスフェクションした後のレポーター遺伝子発現を示します。プラスミドBからのSEAP発現およびプラスミドCからのルシフェラーゼ発現を、カラムベースの手動精製プロセスを用いて、同じ一晩の細菌培養から精製したpDNAのものと比較した。ボックスプロットはデータの分布を示し、各ドットは個々の精製を表す。
結論
この研究では、2.7 kbから11.4 kbまでの4種類のプラスミドを精製し、KingFisher PlasmidPro Maxi Processorの性能を、精製したpDNAの収量やアイソフォーム、トランスフェクション効率、エンドトキシンおよびゲノムDNA含量を含む純度の点で評価しました。KingFisher PlasmidPro Maxi Processorで精製したpDNAの性能と品質は、手動のカラムベースの精製プロセスで達成されたものと同等でした。自動ワークフローで処理された全てのpDNAサンプルは、主にスーパーコイル状pDNAを含み、RNAやgDNAの痕跡は見られず、エンドトキシン含量はそれぞれ0.1 EU/µgをはるかに下回っていました。
これらの結果から、KingFisher PlasmidPro Maxi Processerで単離したpDNAは、タンパク質生産やmRNA合成、ワクチン開発研究、細胞および遺伝子治療に適していることが示されました。
参考文献
1. Stadler J, Lemmens R, Nyhammar T (2004) Plasmid DNA purification. J Gene Med 6(S1):S54–S66. doi:10.1002/jgm.512
2. Lara AR, Ramírez OT (2012) Plasmid DNA production for therapeutic applications. In: Lorence A (editor) Recombinant Gene Expression. Methods in Molecular Biology, vol 824. Totowa, NJ: Humana Press. doi:10.1007/978-1-61779-433-9_14
3. Higgins NP, Vologodskii AV (2015) Topological behavior of plasmid DNA. Microbiol Spectr 3:10.1128/microbiolspec.plas0036-2014. doi:10.1128/microbiolspec. plas-0036-2014
4. Schneier M, Razdan S, Miller AM et al. (2020) Current technologies to endotoxin detection and removal for biopharmaceutical purification. Biotechnol Bioeng 117(8):25882609. doi:10.1002/bit.2736
Ordering information
